这些发现证明了癌症的生长既受到特定癌细胞谱系内在因素的影响,也受到塑造暴露于其中的癌细胞行为的环境因素的影响。
所有生命都连接在一个庞大的族谱树中。每个生物体都与其祖先、后代和旁系亲属存在关系,并且任何两个个体之间的路径都可以被追溯。生物体内的细胞也是如此——人体内数万亿个细胞中的每一个,都是由一个受精卵经过连续分裂产生的,并且可以通过细胞的家族树相互关联。在更简单的生物体(比如秀丽隐杆线虫)中,这种细胞的家族树已被完全绘制出来,但人类的细胞家族树要大得多,也复杂得多。
过去,怀特海德研究所成员 Jonathan Weissman 和其他研究人员开发了谱系追踪(lineage tracing)方法,以跟踪和重建模式生物中细胞分裂的家族树,从而更多地了解细胞之间的关系以及它们如何组装成组织、器官,以及在某种情况下形成肿瘤。这些方法可以帮助回答许多关于生物体如何发育以及像癌症这样的疾病如何起始和进展的问题。

如今,在一项新的研究中,Weissman 及其同事们开发了一种先进的谱系追踪工具,它不仅能够捕获细胞分裂的精确家族树,还将其与空间信息相结合:识别每个细胞最终在组织中的位置。相关研究结果发表在《科学》杂志上。
Weissman 及其实验室博士后研究员Luke Koblan、Kathryn Yost、Pu Zheng 以及研究生 William Colgan 使用他们开发的谱系追踪工具 PEtracer 观察了小鼠体内转移性肿瘤的生长。结合谱系追踪和空间数据,他们能够详细了解癌细胞内在因素及其所处环境因素如何影响肿瘤生长。
Weissman 说道,“理解细胞如何在时间和空间中移动是观察生物学的一个重要方式,在这项新的研究中,我们能够以高分辨率同时看到这两者。其核心思想是,通过了解一个细胞的过去及其最终归宿,你可以看到在其整个生命过程中不同的因素如何影响其行为。”
“在这项研究中,我们使用这些方法来观察肿瘤生长,尽管原则上我们现在可以开始应用这些工具来研究其他感兴趣的生物学问题,比如胚胎发育,” Koblan说。
PEtracer 通过随时间推移在细胞的DNA中反复添加短的、预设的代码来追踪细胞的谱系。每一段代码,称为一个谱系追踪标记(lineage tracing mark),由五个碱基(DNA的构成单元)组成。这些标记使用一种名为先导编辑(prime editing)的基因编辑技术插入,该技术能以最少的非预期副产物直接重写DNA片段。随着时间的推移,每个细胞会获得更多的谱系追踪标记,同时也保留其祖先的标记。然后,该团队可以比较细胞的标记组合,以推断关系并重建家族树。
“我们使用计算建模从第一性原理来设计这个工具,以确保其高度精确,并与成像技术兼容。我们进行了许多模拟,以确定新型谱系追踪工具的最佳参数,然后设计了我们的系统以适应这些参数,” Colgan 说。
当组织——在这项研究中是小鼠肺中生长的肿瘤——充分生长后,该团队收集这些组织,并使用先进的成像方法来观察每个细胞通过谱系追踪标记与其他细胞的谱系关系,以及它在成像组织内的空间位置和其身份(由每个细胞中表达的不同RNA水平确定)。PEtracer 与捕获单细胞遗传信息的成像方法和测序方法都兼容。
“使收集和分析来自成像的所有这些数据成为可能是一个巨大的挑战,” Zheng 说。“让我特别兴奋的不仅是我们能够收集太字节(terabytes)的数据,而是我们设计了这个项目来收集我们知道可以用来回答重要问题并推动生物学发现的数据。”
重建肿瘤的历史
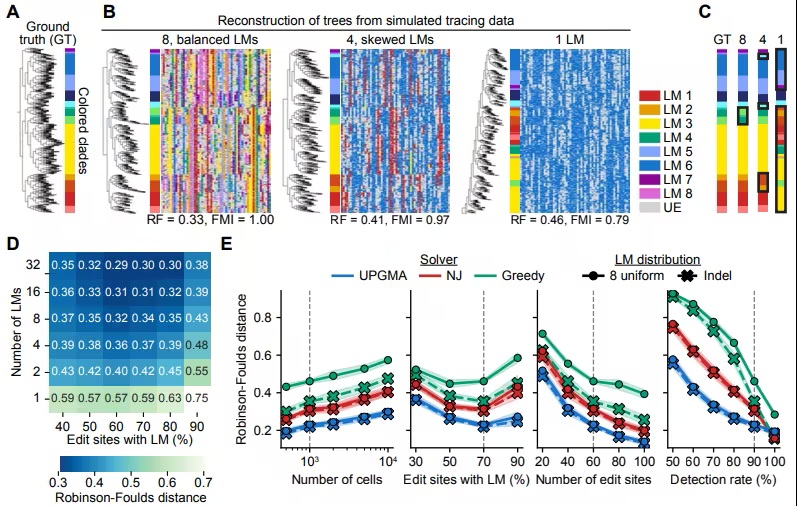
结合谱系追踪、基因表达和空间数据,该团队能够理解这些肿瘤是如何生长的。他们可以判断邻近细胞的亲缘关系有多近,并比较它们的特征。使用这种方法,他们发现他们分析的肿瘤由四个不同的模块或细胞“邻里(neighborhoods)”组成。
最靠近肺部的肿瘤细胞(营养最丰富的区域)适应度(fitness)最高,这意味着它们的谱系历史表明随着时间的推移细胞分裂率最高。癌细胞的适应度往往与肿瘤生长的侵袭性相关。
肿瘤“前沿区(leading edge)”(远离肺部的一侧)的细胞更具多样性且适应度较低。前沿区下方是一个低氧的细胞邻里,这些细胞可能曾经是前沿区细胞,现在被困在一个不太理想的位置。在这些细胞和靠近肺部的细胞之间是肿瘤核心(tumor core),一个包含活细胞、死细胞和细胞碎片的区域。
关键谱系追踪参数的计算机建模
该团队发现,这个家族树上的癌细胞同样有可能最终出现在大多数区域,但靠近肺部的区域除外,因为在那里这个家族树的少数几个分支占据主导地位。这表明癌细胞的不同特征更多地受到其环境(即局部邻里条件)的影响,而不是其家族历史。
证明这一点的进一步证据是,某些与适应度相关的基因(如 Fgf1/Fgfbp1)的表达与细胞的位置相关,而非其祖先。然而,靠近肺部的癌细胞也继承了赋予它们优势的特征,包括表达与适应度相关的基因 Cldn4——这表明家族历史也影响了结果。
这些发现证明了癌症的生长既受到特定癌细胞谱系内在因素的影响,也受到塑造暴露于其中的癌细胞行为的环境因素的影响。
“通过同时观察肿瘤的多个维度,我们获得了在更有限的视角下不可能获得的见解,” Yost 说。“能够表征肿瘤内不同的细胞群体,将使研究人员能够更有效地开发针对最具侵袭性群体的疗法。”
“既然我们已经完成了设计这种工具这项艰巨的工作,我们很兴奋能将其应用于研究健康和疾病、胚胎发育以及其他模式物种中的各种问题,着眼于理解人类健康中的重要问题。我们收集的数据也将有助于训练细胞行为的人工智能模型。我们很期待与其他研究人员分享这项技术,看看我们能共同发现什么,” Koblan 说。
这种新开发的AI平台旨在解决癌症免疫疗法中的一个主要挑战:展示科学家如何开发针对肿瘤细胞的靶向治疗,同时避免损伤健康组织。
在一项新的研究中,来自丹麦技术大学和斯克里普斯研究所的研究人员开发出一种人工智能(AI)平台,能够定制蛋白质组件,从而武装患者的免疫细胞来对抗癌症,这使得更大规模的精准癌症治疗离现实更近了一步。相关研究结果发表在《科学》杂志上。

这种新方法首次证明,可以在计算机上设计蛋白质,通过pMHC分子将免疫细胞重定向以靶向癌细胞。这将寻找有效抗癌分子的过程从数年戏剧性地缩短至几周。
“我们本质上是在为免疫系统创造一双新的眼睛。目前个体化癌症治疗的方法是基于在患者或供者的免疫系统中寻找可用于治疗的所谓T细胞受体(T-cell receptors)。这是一个非常耗时且具有挑战性的过程。我们的平台利用AI平台设计靶向癌细胞的分子‘钥匙’,其速度令人难以置信,因此一个新的先导分子可以在4-6周内准备就绪,”丹麦技术大学副教授、论文共同通讯作者 Timothy P. Jenkins 解释道。
对抗癌症的“精确制导导弹”
这种新开发的AI平台旨在解决癌症免疫疗法中的一个主要挑战:展示科学家如何开发针对肿瘤细胞的靶向治疗,同时避免损伤健康组织。
通常情况下,T细胞通过识别由pMHC分子呈现在细胞表面的特定蛋白质片段(称为肽)来自然识别癌细胞。将这一知识应用于治疗是一个缓慢且具有挑战性的过程,通常是因为人体自身T细胞受体的多样性使得创建个性化治疗变得困难。
增强人体免疫系统
在该研究中,研究人员在一个众所周知的癌症靶点NY-ESO-1上测试了这种AI平台的能力,其中该靶点存在于多种癌症中。他们成功设计了一种能与NY-ESO-1 pMHC分子紧密结合的微型结合蛋白(minibinder)。当将这种设计蛋白质(deisigner protein)插入T细胞时,它创造了一种独特的新型细胞产品,他们将其命名为IMPAC-T细胞。在实验室实验中,它们有效地引导T细胞杀死了癌细胞。
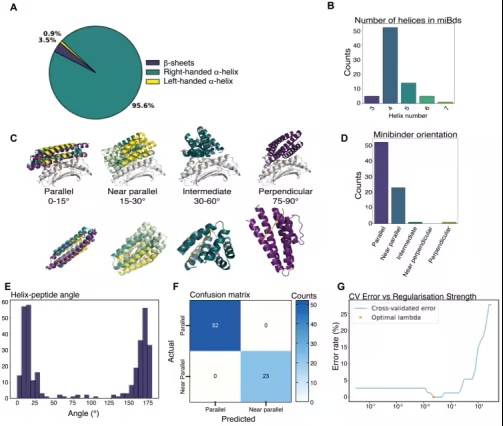
靶向SLLMWITQC/HLAA*02:01 NY-ESO-1 pMHC的miBds的结构与定位分析
“将完全在计算机上创建的这些微型结合蛋白拿到实验室,并看到它们如此有效地发挥作用,这令人无比兴奋,”论文共同第一作者、丹麦技术大学研究员 Kristoffer Haurum Johansen说道。
研究人员还利用这种平台设计出靶向一种在一名转移性黑色素瘤患者体内发现的癌症靶点的微型结合蛋白,并成功为该靶点生成了微型结合蛋白。这证明了该方法也可用于针对新型癌症靶点的定制化免疫治疗。
治疗方案的筛选
研究人员这一创新发现中的一个关键步骤是开发了一种虚拟安全筛查(virtual safety check)。他们利用AI平台对他们设计的微型结合蛋白进行筛选,并评估它们与健康细胞上发现的pMHC分子的关系。这种方法使他们能够在进行任何实验之前,就过滤掉可能导致危险副作用的微型结合蛋白。
“癌症治疗的精准性至关重要。通过在设计阶段就预测并排除交叉反应(cross-reactions),我们能够降低设计蛋白相关的风险,并增加设计出安全有效疗法的可能性,”丹麦技术大学教授、论文共同通讯作者 Sine Reker Hadrup 表示。
五年内进入治疗阶段
Jenkins预计,这项新方法需要长达五年的时间才能准备好进行首次人体临床试验。一旦该方法成熟,治疗过程将类似于目前使用经过基因修饰的T细胞(即CAR-T细胞)的癌症治疗方法(目前用于治疗淋巴瘤和白血病)。
具体过程就是患者将首先在医院抽血,类似于常规血液检查;然后,从这份血液样本中提取他们的T细胞,并在实验室进行改造,使其携带AI平台设计的微型结合蛋白。这些增强的T细胞随后被回输给患者,它们就像精确制导导弹一样,在体内精准地寻找并消灭癌细胞。
本研究对CRBN(Cereblon)靶点空间的系统性探索揭示了CRBN的非凡可塑性,重新定义了靶点结合规则,并为在β-发夹G-环范式内外的靶点空间扩展提供了机遇。
分子胶(molecular glue),即能够将一种蛋白质与另一种蛋白质连接起来的微小分子,是药物研究中极具潜力的靶点。通过将与一种疾病相关的蛋白质与一种触发细胞降解和回收通路的蛋白质相连,药物研究人员成功开发出针对药物耐药性疾病的新型疗法。然而,人们曾认为这种药物开发方法仅限于那些具有特定表面特征——β-发夹环基序(beta-hairpin loop motif)的蛋白质。

在这个曾经狭窄的发现空间上,一项新的研究揭示了一系列能够与分子胶降解剂(一种分子胶分子的药用版本)结合的蛋白质表面特征。这些结果可能通过靶向以前被认为对药物治疗“不可触及”的蛋白质,为治疗疾病开辟新的途径。相关研究成果发表于《科学》杂志。
分子胶降解剂的作用机制是首先与参与细胞蛋白质回收过程的蛋白质Cereblon结合。随后,分子胶降解剂在Cereblon蛋白质表面引发微小变化,实质上为原本无法访问的目标蛋白质创建了一个停泊位点。一旦结合在一起,Cereblon用一种叫做E3泛素连接酶的分子标记这种目标蛋白质,从而触发细胞分解该蛋白质。
研究人员首先通过计算方法在人类蛋白质组中搜索可能与一种典型分子胶受体匹配的特征。这一过程最初发现了1633种人类蛋白质,由于其表面存在环状结构,可能与Cereblon兼容。这种挖掘方法不仅在人类蛋白质组中发现了β-发夹环,还发现了螺旋环,后者是一种结构上不同但足够兼容的识别基序。
在他们的发现中,研究人员识别出VAV1,即一种此前在自身免疫性疾病和慢性炎症疾病中具有广泛治疗潜力的药物无法接触到的蛋白质。
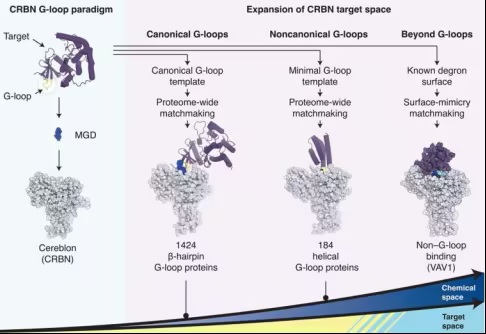
研究人员写道,“我们对CRBN(Cereblon)靶点空间的系统性探索揭示了CRBN的非凡可塑性,重新定义了靶点结合规则,并为在β-发夹G-环范式内外的靶点空间扩展提供了机遇。”
值得注意的是,药物沙利度胺(thalidomide)后来被发现是一种分子胶降解剂。尽管因其在怀孕期间服用时导致的严重副作用而在20世纪60年代被撤出市场,但由于其治疗某种癌症的能力,它后来重新受到关注,并成为治疗汉森病(Hansen's disease)患者炎症的有效疗法。
文章转载自生物谷,系出于传递更多信息之目的,转载内容不代表本站立场。如有侵权请及时联系,我们将立即进行删除处理。




 京公网安备 11011402010692号
京公网安备 11011402010692号




